Introduction
Lire des tableaux de chiffres peut etre long et fastidieux, alors qu’il est bien plus facile et rapide de comparer visuellement des longueurs de barres ou des degrades de couleurs. Dans les paragraphes qui suivent, on a un apercu des possibilites offertes par R et Python, pour des mises en forme generales ou plus specialisees comme les cartes de chaleur.
library("reactable")
library("glue")
library("heatmaply")
library("RColorBrewer")
# jeux de données
donnees = mtcars
donnees$mpg = donnees$mpg - mean(donnees$mpg)
donnees$drat = donnees$drat - mean(donnees$drat)
url = "https://archive.ics.uci.edu/ml/machine-learning-databases/wine-quality/winequality-white.csv"
wine_quality = read.csv(url, sep = ";")import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from scipy.cluster.hierarchy import linkage
from scipy.spatial.distance import pdist
sns.set()
donnees = r.donnees
wine_quality = r.wine_quality
mtcars = r.mtcarsTables numeriques generales
dans Python avec pandas
On a une syntaxe tres compacte et efficace sous pandas.
html = (donnees[['mpg', 'drat', 'disp', 'cyl', 'hp']].iloc[:10].style
.set_table_styles([{'selector':"tr:hover", 'props':[("background-color", "#ffff99")]}])
.set_properties(**{'font-size': '9pt', 'font-family': 'Calibri','padding-right': '5px',\
'border': '1px solid grey'})
.set_precision(2)
.applymap(subset = 'mpg', func = lambda x: f"color: {'red' if x < 0 else 'blue'}")
.bar(subset = 'drat', color = ['coral', 'lightblue'], align = 'zero')
.highlight_max(subset = 'cyl', color = 'lightgreen')
.highlight_min(subset = 'cyl', color = 'orange')
.background_gradient(subset = 'hp', cmap = 'Blues')
.render()
)
# with open('datatable.html','w') as f:
# f.write(html)| mpg | drat | disp | cyl | hp | |
|---|---|---|---|---|---|
| Mazda RX4 | 0.91 | 0.30 | 160.00 | 6.00 | 110.00 |
| Mazda RX4 Wag | 0.91 | 0.30 | 160.00 | 6.00 | 110.00 |
| Datsun 710 | 2.71 | 0.25 | 108.00 | 4.00 | 93.00 |
| Hornet 4 Drive | 1.31 | -0.52 | 258.00 | 6.00 | 110.00 |
| Hornet Sportabout | -1.39 | -0.45 | 360.00 | 8.00 | 175.00 |
| Valiant | -1.99 | -0.84 | 225.00 | 6.00 | 105.00 |
| Duster 360 | -5.79 | -0.39 | 360.00 | 8.00 | 245.00 |
| Merc 240D | 4.31 | 0.09 | 146.70 | 4.00 | 62.00 |
| Merc 230 | 2.71 | 0.32 | 140.80 | 4.00 | 95.00 |
| Merc 280 | -0.89 | 0.32 | 167.60 | 6.00 | 123.00 |
dans R avec reactable
En utilisant le package formattable le code serait beaucoup plus court, mais il ne permettrait pas de faire des barres de couleurs differentes pour les valeurs positives ou negatives. Le package reactable offre beaucoup plus de possibilites au prix d’un code plus long, cf quelques hebdomadR precedents sur le format des tables.
ma_table = donnees[1:10, c('mpg', 'drat', 'disp', 'cyl', 'hp')]
largeur_colonnes = 100
# fonction pour avoir des barres positives et negatives
bar_style <- function(width, height = "60%") {
new_width = round(50 * (width + 1))
if (width > 0) {
image = glue("linear-gradient(90deg, transparent 0%, 50%, lightblue 50%, {new_width}%,
transparent {new_width}%)")
} else {
image = glue("linear-gradient(90deg, transparent 0%, {new_width}%, coral {new_width}%,
50%, transparent 50%")
}
list(backgroundImage = image,
backgroundSize = paste("100%", height),
backgroundRepeat = "no-repeat",
backgroundPosition = "center",
color = "black")
}
# fonction pour la palette de couleur Blues
blue_pal <- function(x) rgb(colorRamp(brewer.pal(9,"Blues"))(x),
maxColorValue = 255)
reactable(
ma_table,
bordered = TRUE,
highlight = TRUE,
columns = list(
disp = colDef(width = largeur_colonnes),
mpg = colDef(style = function(value) {
if (value < 0) {color = "red"} else {color = "blue"}
list(color = color)},
format = colFormat(digits = 2),
width = largeur_colonnes),
cyl = colDef(style = function(value) {
color = "none"
if (value == max(ma_table$cyl)) {color = "lightgreen"}
if (value == min(ma_table$cyl)) {color = "orange"}
list(background = color)},
width = largeur_colonnes),
hp = colDef(style = function(value) {
normalized <- (value - min(ma_table$hp)) / (max(ma_table$hp) - min(ma_table$hp))
list(background = blue_pal(normalized))},
width = largeur_colonnes),
drat = colDef(style = function(value) {bar_style(width = value / max(abs(ma_table$drat)))},
align = "center",
format = colFormat(digits = 2),
width = largeur_colonnes))
)Cartes de chaleur
Que ce soit sous R ou Python, les cartes de chaleur operent par defaut un tri intelligent par CAH sur les lignes et colonnes. Et les 2 packages choisis utilisent ici la palette viridis.
dans Python avec seaborn
g = sns.clustermap(mtcars, figsize=(5, 5), z_score = 1, cmap = "viridis")
plt.show()
dans R avec heatmaply
heatmaply(mtcars, scale = "column", plot_method ="plotly")Matrices de correlation
On veut que 2 colonnes tres correlees ou anti-correlees soient proches, on utilise pour cela la mesure de similarite \(1 - \text{abs}(\text{cor}(X,Y))\).
dans Python avec seaborn
On utilise le clustering par CAH pour trier intelligemment les colonnes de la matrice de correlation, puis on la visualise par carte de chaleur.
# tri intelligent des colonnes
link = linkage(1 - np.abs(1 - pdist(wine_quality.T, 'correlation')), 'ward')
clustergrid = sns.clustermap(wine_quality, col_linkage = link, row_cluster = False, figsize=(5, 5))
plt.show()
# on applique le nouvel ordre
ordre_col = list(wine_quality.iloc[:,clustergrid.dendrogram_col.reordered_ind].columns)
mat_corr = wine_quality[ordre_col].corr()
plt.figure(figsize = (11,11))
sns.heatmap(mat_corr, vmin = -1, vmax = 1, center= 0, annot = True, cmap = "RdBu")
plt.show()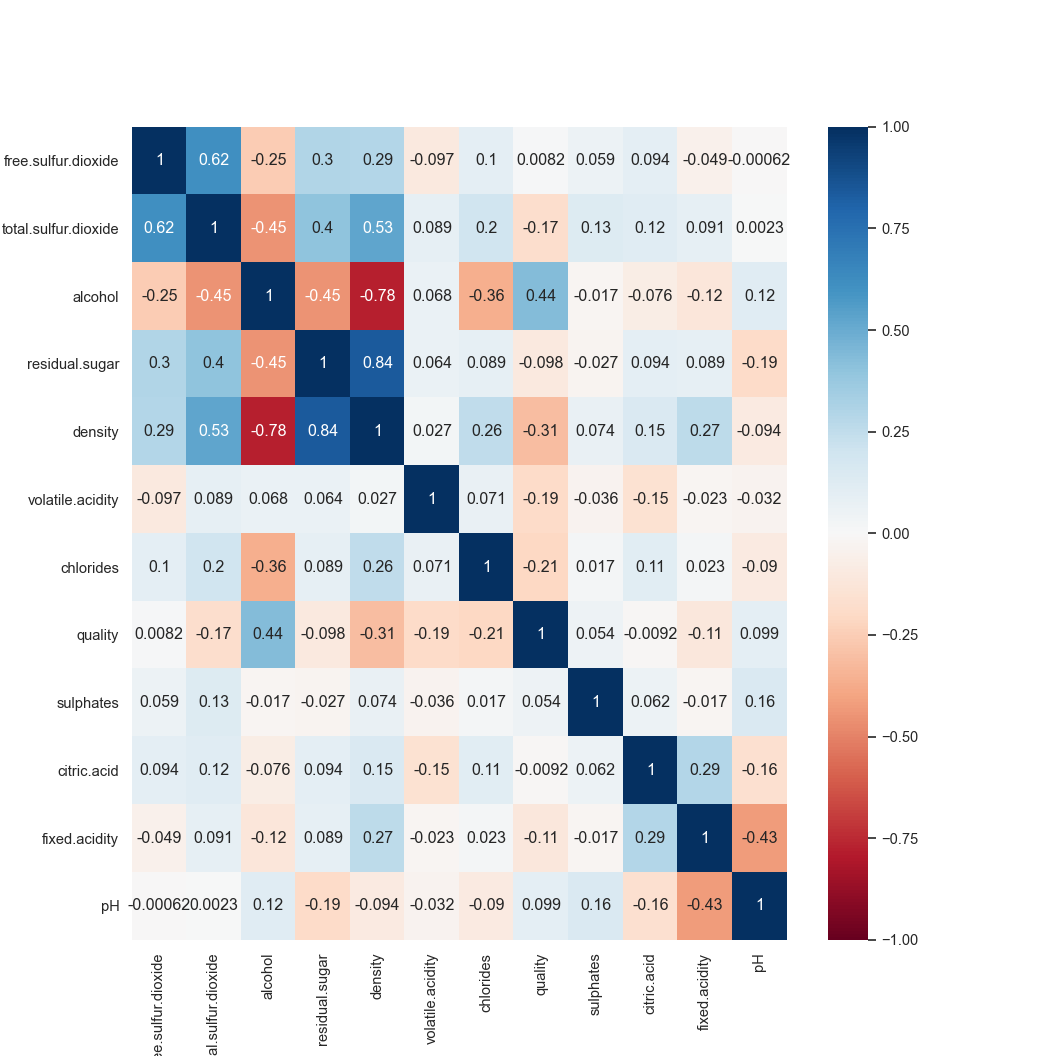
dans R avec heatmaply
C’est la carte de chaleur qui effectue la CAH, et on retrouve les memes groupes de variables que sous Python.
ma_dist = function(x) {as.dist(1 - abs(cor(t(x))))}
mat_corr = cor(wine_quality)
heatmaply(mat_corr,
distfun = ma_dist, hclust_method = "ward.D",
colors = c("red", "white", "blue"), limits = c(-1, 1),
revC = FALSE, symm = TRUE,
cellnote = round(mat_corr, 2), cellnote_textposition = "middle center",
plot_method = "plotly") %>% plotly::layout(width=900)